怎么构建电脑系统硬盘-怎么构建电脑系统
1.群体结构——系统发育树的构建
2.为什么用Linux内核来构建嵌入式操作系统?
群体结构——系统发育树的构建
最近杂事真的非常的满,终于找到时间更新一下。。。。
通过上一篇文章的介绍, 系统发育树的基本概念 大家已经了解清楚,那到底怎么获得一棵可信的进化树呢?
对于群体遗传学分析,一般都会以群体SNPs位点数据构建系统发育树,因此,接下来我主要以SNPs数据为例,介绍系统进化树的构建方法。
序列比对->建树方法选择->计算最佳替代模型->进化树建立->进化树美化
常见的序列比对软件包括:Clustal和Muscle等。
Clustal 除了有自己独立的软件外(多种操作系统都支持),也常被整合到一些常见的软件中,如:Bioedit、MEGA等。
Muscle 同样支持多种操作系统。
两个软件的引用频率都很高,没有绝对的谁好谁坏,哪个顺手就用哪个即可。
1、Distance-based methods 距离法:
基于距离的方法:首先通过各个物种之间的比较,根据一定的假设(进化距离模型)推导得出分类群之间的进化距离,构建一个进化距离矩阵。进化树的构建则是基于这个矩阵中的进化距离关系。
2、Character-based methods 特征法:
基于特征的方法:不计算序列间的距离,而是将序列中有差异的位点作为单独的特征,并根据这些特征来建树。
模型选择的依据如下图:
UPGMA法已经较少使用。一般来讲,如果模型合适,ML的效果较好。对近缘序列,有人喜欢MP,因为用的假设最少。MP一般不用在远缘序列上,这时一般用NJ或ML。对相似度很低的序列,NJ往往会出现Long-branch attraction(LBA,长枝吸引现象),有时严重干扰进化树的构建。贝叶斯方法则太慢。对于各种方法构建分子进化树的准确性,有一篇综述 (Hall BG, 2005) 认为贝叶斯的方法最好,其次是ML,然后是MP。其实如果序列的相似性较高,各种方法都会得到不错的结果,模型间的差别也不大。不过现在文章普遍使用的是NJ是ML模型。
系统发育分析中,最大似然法(ML)和贝叶斯法(BI)是对替代模型非常敏感的两种算法,因此,利用ML法或BI法重建系统发育树前,替代模型的选择是必不可少的过程。
Win操作系统下jModeltest的使用方法参考这篇文章: 图解核苷酸替代模型的选择 - jModelTest 篇(By Raindy) 。
ProTest的使用方法可以参考这篇文章: 使用 ProtTest 来选择最优氨基酸替代模型 。
我自己基本都用的是Linux版本的jModelTest,使用及其简单,命令如下:
参数说明:
-d:输入文件。注意!这个软件需要输入的是.phy格式文件,不是.fasta格式。
-f:include models with unequals base frecuencies
-g:include models with rate variation among sites and number of categories
-i: include models with a proportion invariable sites
-s:number of substitution schemes
-v:do model averaging and parameter importances
-a:estimate model-averaged phylogeny for each active criterion
-BIC:calculate the Bayesian Information Criterion
-AIC:calculate the Akaike Information Criterion
结果的最下方,有如图所示的列举,也就是得分最高的模型。
计算完最佳模型,我们就要开始建树了。对于ML树的构建,推荐大家使用新一代RAxML——raxml-ng。
RAxML一直是ML建树的经典工具,其由来自德国海德堡理论科学研究所(Heidelberg Institute for Theoretical Studies)的Alexandros Stamatakis开发。近年来,其江湖地位也受到来自其他软件,尤其是IQ-Tree的挑战。Zhou等人的文章 Evaluating Fast Maximum Likelihood-Based Phylogenetic Programs Using Empirical Phylogenomic Data set 对RAxML,IQ-TREE,FastTree,Phyml四个最大似然法建树软件的实际效果和表现进行了系统比较,其中一个结论是IQTREE在准确性方面要略胜一筹。
近日,RAxML的升级版, raxml-ng 发布!
相较于上一代,raxml-ng有如下优势:
话不多说,直接建树:
参数说明:
--all:Perform an all-in-one analysis (ML tree search + non-parametric bootstrap)
--msa:对其后的序列文件
--model:直接输入上一步产生的最佳模型
--bs-trees:检查树的鲁棒性(robustness)进行自展(bootstrap)检验,进行1000次bootstrapping抽样
--threads:给定线程
运行后结果如下图所示,其中.bestTree就是我们要的树文件,导入树可视化工具即可(我比较常用MEGA和 iTOL ),下次再写一下如何美化进化树吧。
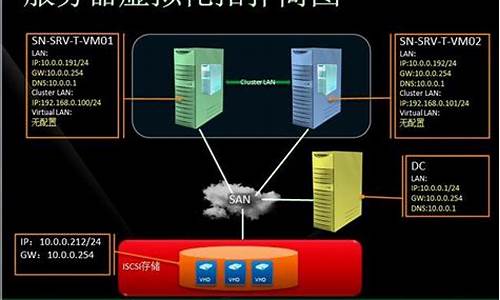
做进化分析的工友们可能都有个感觉,很多分析一等就是好几天,特别是建树(做过的都知道其中的痛苦),有时候忽然加入一个样品又要从头来。因此,一台给力的服务器是必要的工具。比如,上文提到了SNP进化树,我做的还仅仅只是相近物种,而且基因组很小(9M),SNP位点就有4万个,如果要用我MEGA这些软件调用我电脑8核的CPU,1000自展值可能要跑到毕业。
生物学背景出身的我,抄着那一点可怜的计算机常识,在我们课题组购买服务器时,我做了非常多的功课。当然,主要还是听取公司技术人员的建议,通过我非常非常非常长时间的测试,多次使用常见的生物信息分析软件(我主要从事寄生虫基因组、宿主转录组、16S宏基因组等研究),最终,找到了一个性价比超高的服务器配置,具体配置如下:
真心感谢一下烽伟的技术小哥哥们,乐不疲的回答我各种低级的问题,如果有啥需要可以联系一下他们的技术,感觉蛮靠谱哒,官网: 烽伟科技 。
上一个他们的LOGO,以表感谢。
本文为本人的学习笔记,希望对大家有所帮助。本文大量参考网络文章,文章来源列举于全文末尾。
参考:
一文读懂进化树
使用 ProtTest 来选择最优氨基酸替代模型
RAxML进化树构建的新一代——raxml-ng
为什么用Linux内核来构建嵌入式操作系统?
这个问题应该类似于linux内核构建嵌入式操作系统有什么优势呢?
嵌入式系统的商品化操作系统十分丰富,如Palm OS、VxWorks、pSOS、Neculeus和Windows CE等.高端嵌入式系统需要许多高级的功能,但其价格也相对昂贵,一般用户难以接受.微软的Windows CE也有此类功能,却不具备大多数嵌入式系统需要的实时性能,而且难以移植.
Linux为嵌入式系统提供了一个极有吸引力的选择,对于嵌入式系统而言,性能、成本和可靠性是最至关重要的三个因素.首先,众多文献资料表明,Linux是当前可获得的最简捷、最快速的操作系统 ,其性能优越之处,是把图像处理为一个用户级的应用,图像可根据需要被选择是否运行.Linux系统中存在适度复杂的图像界面,但是他们并没有和操作系统的内核紧紧捆绑在一起,图像界面可按需求关闭.这样就能够在Linux内核上运行专门为嵌入式系统定制的图像系统,从而获得优越的性能.其次,Linux 系统源代码完全公开,能够用很便宜的价格得到各种Linux分发版,不必考虑许可成本,将用户从许可证的限制中解脱出来,无需去为资金短缺而烦忧.Linux能正常运行于内存缺乏,容量紧张的系统中,减少在硬件升级上的开支.另外,在系统稳定性方面,Linux几乎不崩溃,Linux的稳定性是由于他没有像其他操作系统相同内核极其庞大.考察资料表明,Linux和其他Unix系统和大型操作系统如VMS、IBM大型机等相同具备相同的可靠性. 在上述优势之外,Linux还拥有众多硬件支持的特点和强大的网络支持功能.正因为Linux在价格、性能、稳定性连同用户定制等方面的突出优势,用他来构建系统操作平台是个很不错的解决方案
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。